大学院理工学研究科の平田章講師、西山聖示さん、田村俊浩さん、山内綾乃さん、堀弘幸教授は、太古の生命体に近い超好熱菌を用い、tRNA分子にメチル化を施す責任酵素aTrm11の基質認識機構について、構造生物学的観点から説明することに成功しました。aTrm11はS-アデノシルメチオニンをメチル基供与体として利用し、tRNAにおける10番目のG(G10)のN2-メチルグアノシン (m2G10)およびN2、N2-ジメチルグアノシン(m22G10)形成を触媒します。aTrm11が発見されて12年経過しても、aTrm11の分子構造情報が利用できず、その詳細な基質認識メカニズムは不明でした。
そこで、我々は、超好熱性微生物Thermococcus kodakarensis由来のaTrm11を結晶化し、大型放射光施設 (SPring-8)のX線を利用して、aTrm11のX線結晶構造を決定しました(図1)。aTrm11は主に3つのパーツ(NFLD、THUMP、RFM)で構成され、THUMPとRFMが1つのリンカーで繋がれていました。aTrm11の分子構造を基盤に生化学的解析を行った結果、aTrm11のTHUMP部分がtRNA中の3’-CCA末端の1本鎖を認識し、他のパーツもtRNA認識に重要であることが明らかになりました(図2)。
また、類縁のtRNAメチル化酵素と比較した結果、RFMパーツの空間的配置がtRNA中のどの部位のGをメチル化するか(部位決定)に寄与していることを推定しました。本研究により、酵素の絶妙な「かたち」が「働き」に依存するという構造機能相関の一端を解明したと言えます。
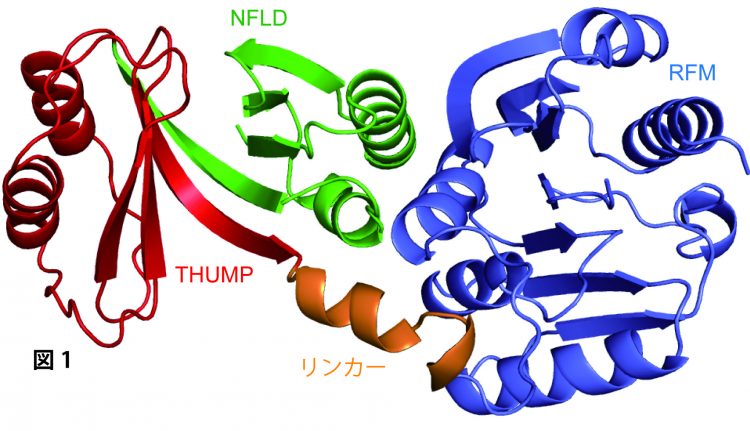
図1 aTrm11 のX線結晶構造のリボンモデル図。緑色はNFLD (N-terminal feredoxin-like domain)、赤色はTHUMP (THioUridine synthase, Mthyltransferase, and Pseudouridine synthase)、薄青色はRFM (Rossmann-fold methyltransferase)、橙色は リンカー領域をそれぞれ示している。
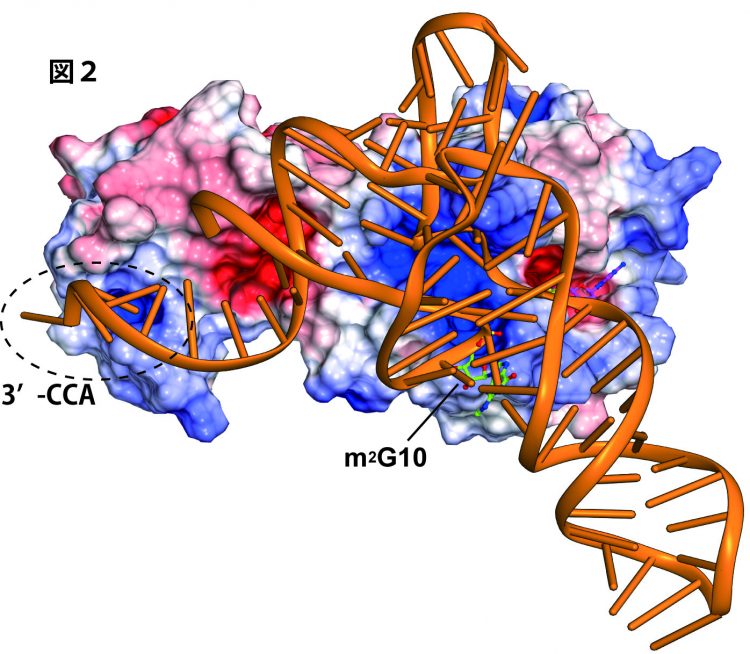
図2 aTrm11と基質tRNA複合体の推定モデル図。aTrm11は静電ポテンシャル表面分布図で示し、負電荷を赤色、正電荷を青色で表示している。tRNAの3’-CCA末端配列をTHUMPが認識しているのが特徴的であり、正電荷の部分がtRNAの認識に重要である。
本研究成果は、核酸化学の分野でトップレベルにある英国科学雑誌「Nucleic Acids Research」に平成28年6月20日にオンライン掲載されました。
掲載先URL:http://nar.oxfordjournals.org/cgi/content/full/gkw561?ijkey=RIFYBIjVm3JcNz5&keytype=ref
なお、本研究は日本学術振興会科学研究費および愛媛大学研究活性化事業「RNA科学の拠点形成」による支援を受けました。
また、愛媛大学学術支援センター物質科学部門および応用タンパク質研究部門における共同機器利用の支援も受けました。共著者一同、深く感謝申し上げます。
最後に、当時、大学院理工学研究科修士課程2年生だった田村俊浩さんが、aTrm11の結晶を作成することに成功したため、本研究を遂行することができました。残念ながら、田村さんは2013年3月に逝去したため、aTrm11の分子構造を見ることは叶いませんでした。しかし、今日ここに研究成果を報告できたことに彼の遺志を感じてなりません。共著者一同、謹んで田村さんのご冥福をお祈り申し上げます。

